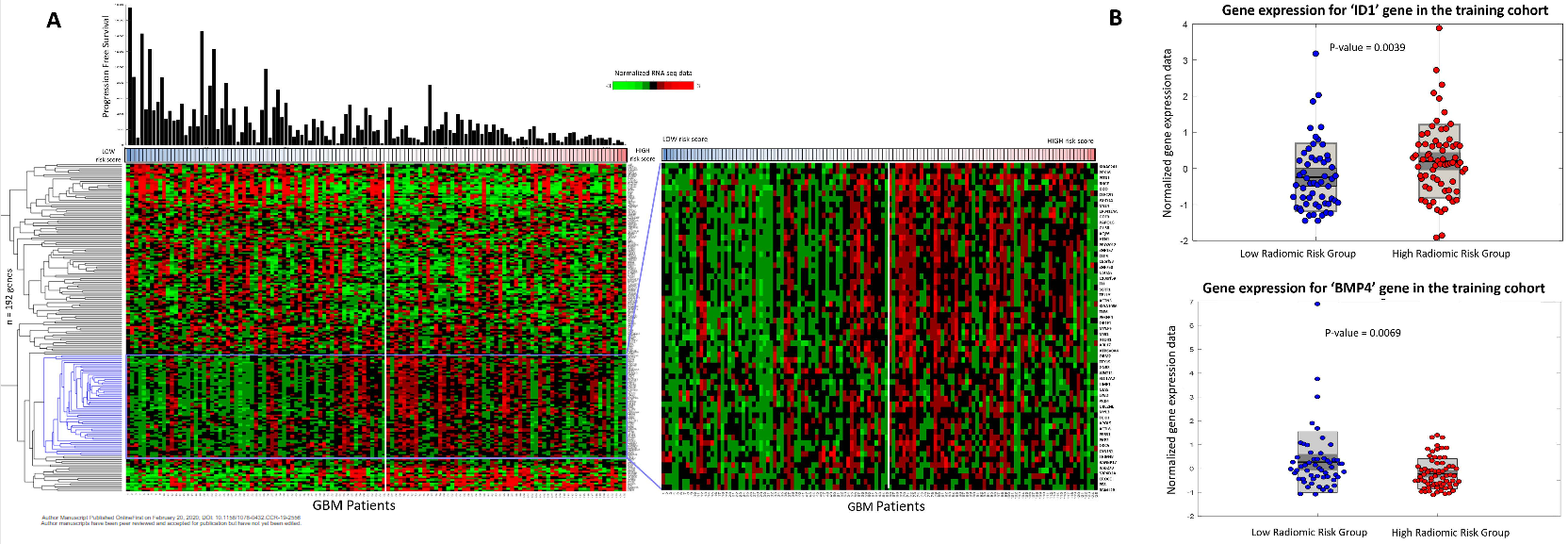
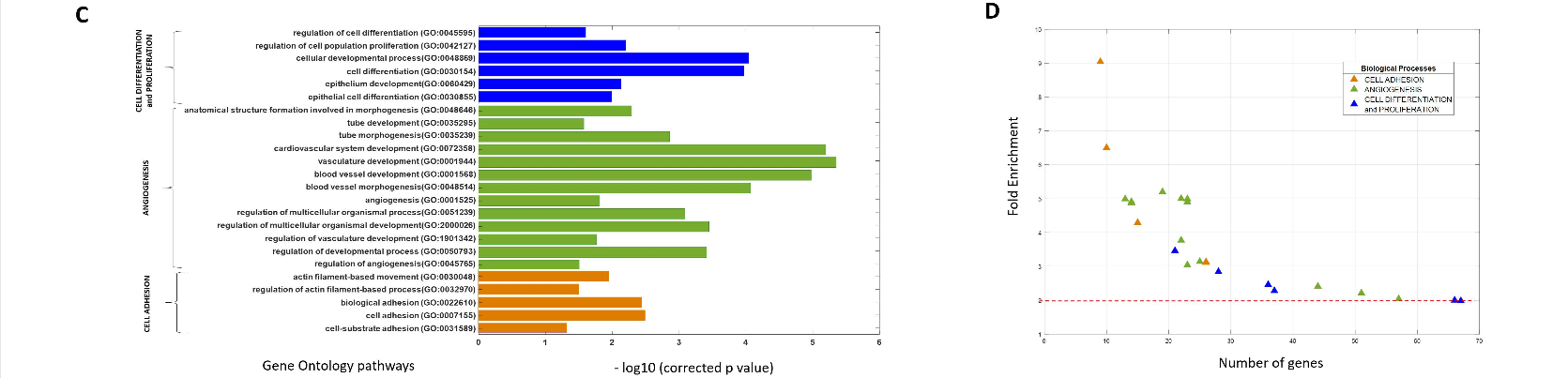
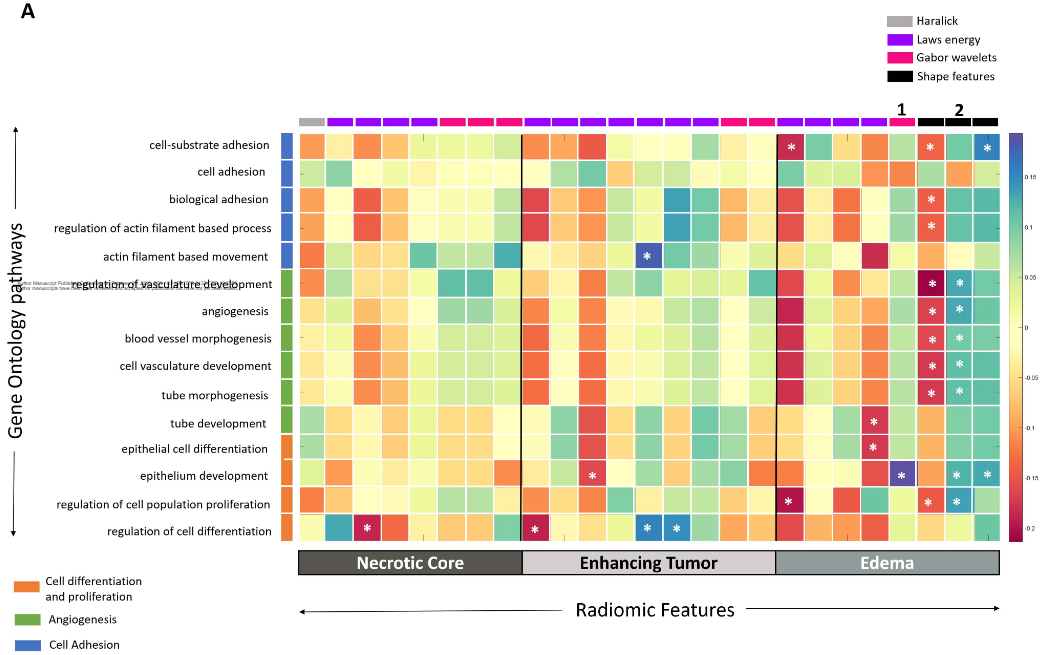
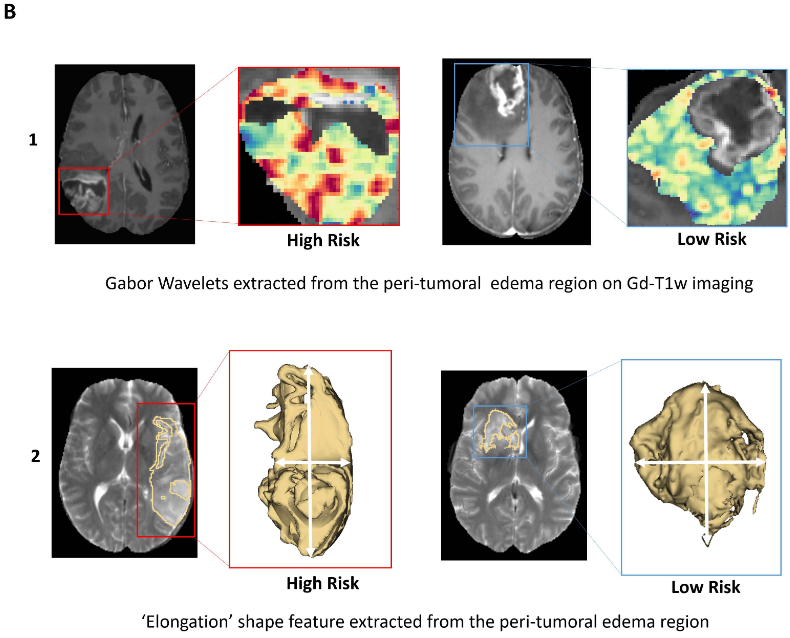
美国俄亥俄州克利夫兰凯斯西储大学的Niha Beig等研究胶质母细胞瘤患者的影像组学与基因组学,从MRI-T1加权增强序列中提取与患者无进展生存期(PFS)密切相关的影像学特征,进行生物信息学分析,发现影像学特征与细胞粘附、细胞增殖、细胞分化和血管生成等胶质瘤耐药核心过程密切相关,认为未来通过影像组学的深度分析,可实现无创性胶质瘤分子病理诊断。文章发表于2020年2月的《Clinical Cancer Research》在线。
——摘自文章章节
研究背景
研究方法
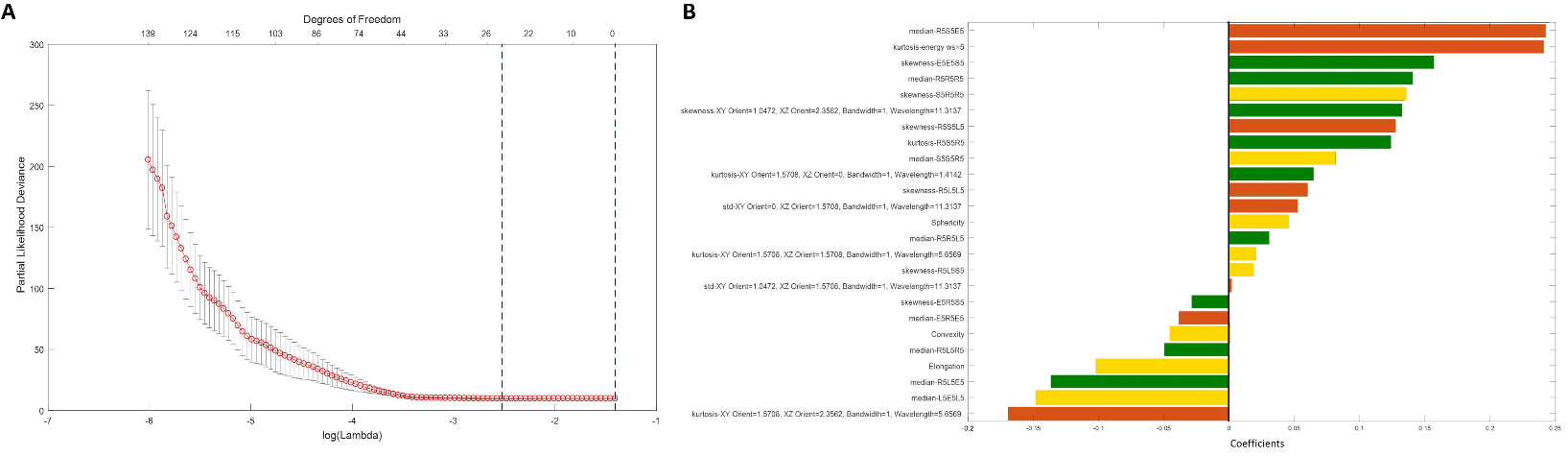
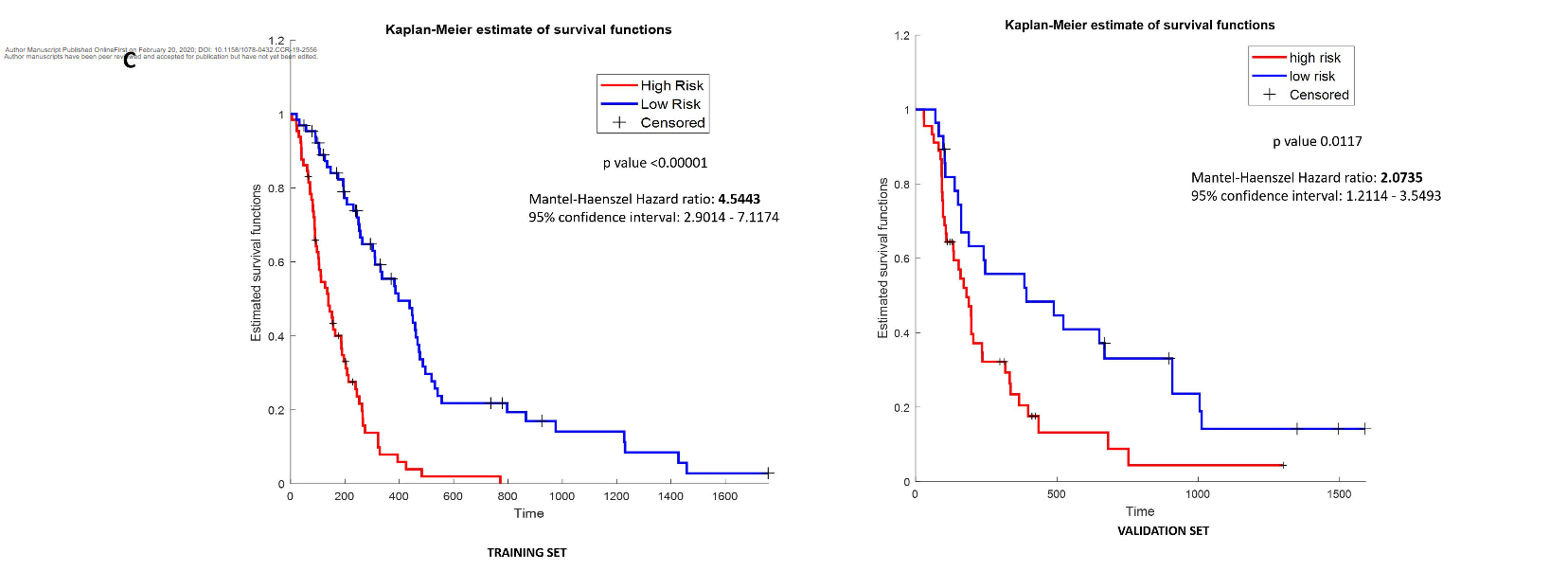
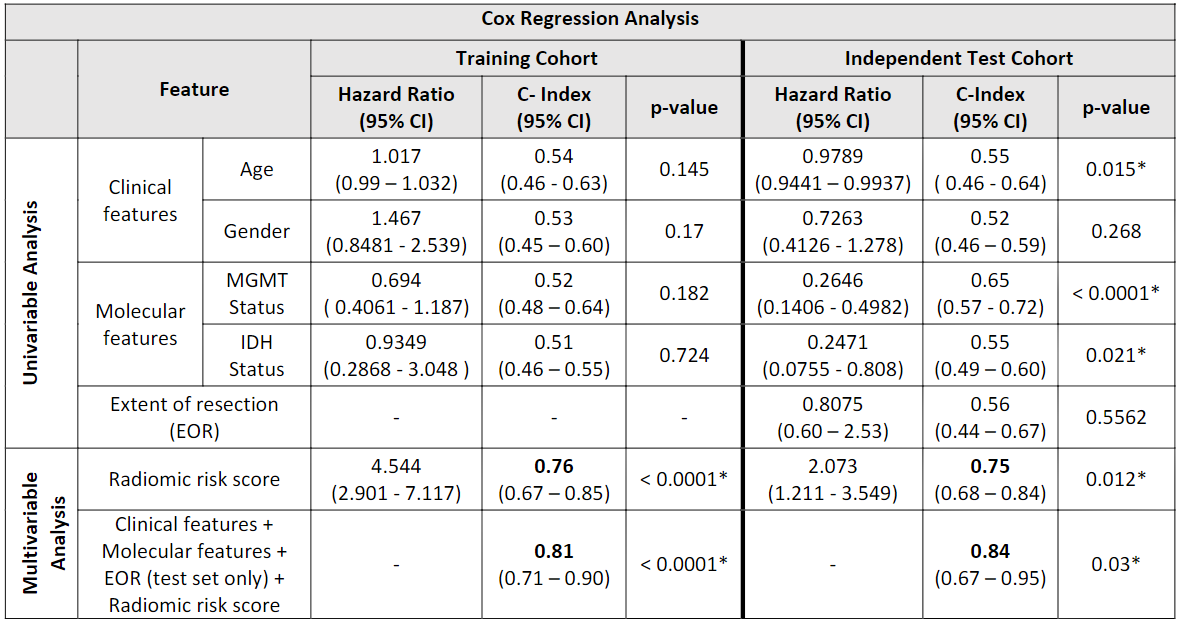
研究结果
结论
综上所述,该研究结果表明,常规MRI-T1加权增强所发现的、与预后相关的影像组学特征与GBM治疗耐药性核心生物学过程密切相关。






